折線圖是將散布圖上的資料點,用線條連接起來的一種圖形,目的是觀察資料分布的趨勢,特別是用在時間序列上。折線圖的原始型態就是點圖,因此在R Base裡的繪圖指令是用plot()並搭配type="p"、type="l"、type="b"等參數來做點圖、線圖或點線圖的變化。geom_line()則是ggplot2裡的折線圖繪圖指令。
折線圖
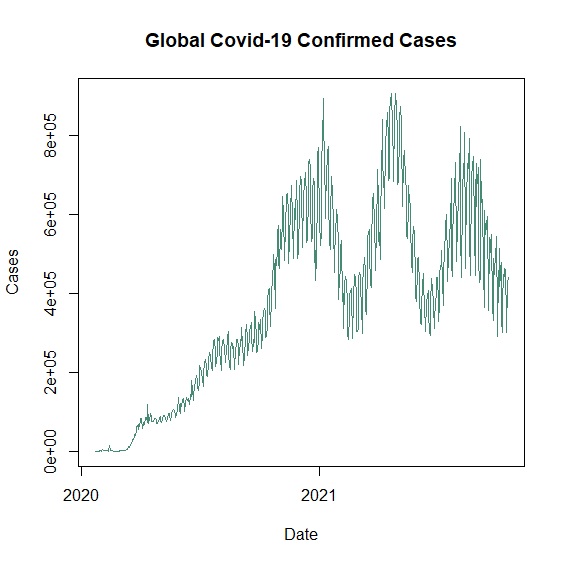
一個簡單的折線圖,在plot()設定好x軸與y軸後就可以畫出來。這裡以covid19.csv為例畫出全球的新冠肺炎確診人數趨勢圖,因為資料裡的Date是字串,首先要將Date由原本的字串改成日期。
> covid<-read.csv("c:/Users/USER/downloads/covid19.csv", header=T, sep=",")
> covid[,c("Date")]<-as.Date(covid[,c("Date")])
> attach(covid)
> plot(x=Date, y=World, type="l", xlab="Date", ylab="Cases", main="Global Covid-19 Confirmed Cases", col="aquamarine4")
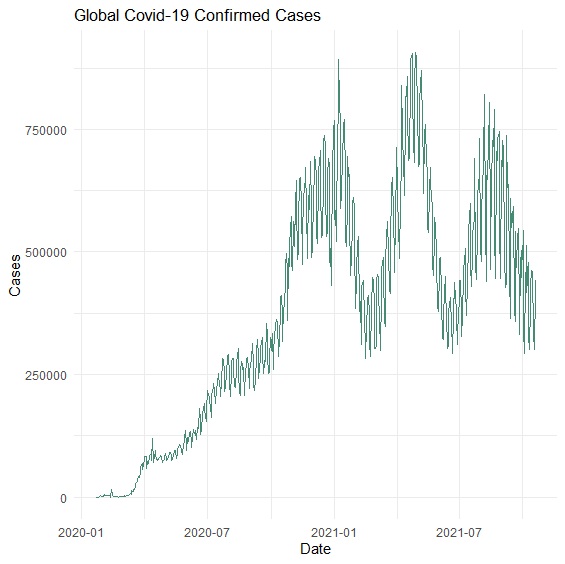
ggplot折線圖指令是geom_line()。
> p<-ggplot(covid, aes(x=Date, y=World))+
+ geom_line(color="aquamarine4")+
+ ggtitle("Global Covid-19 Confirmed Cases")+
+ labs(x="Date", y="Cases")+
+ theme_minimal()
> p
疊合折線圖
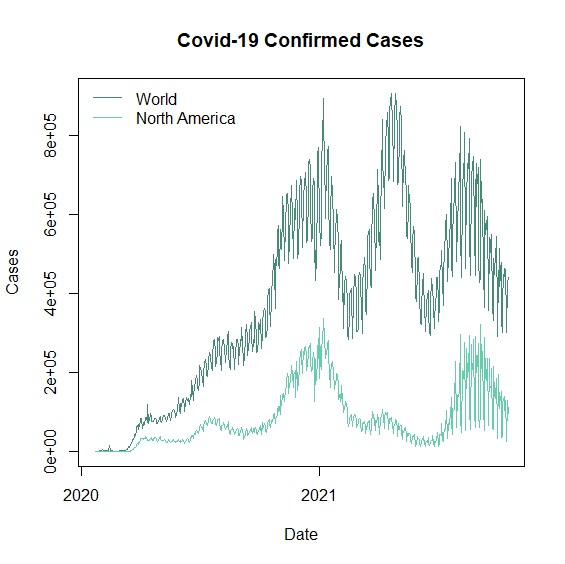
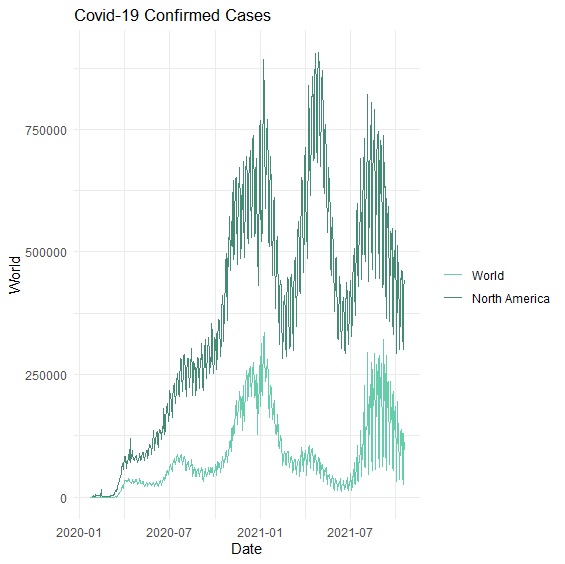
有多種線條要呈現在同一張圖的時候,R Base只需要用lines()指令。
> plot(x=Date, y=World, type="l", xlab="Date", ylab="Cases", main="Covid-19 Confirmed Cases", col="aquamarine4")
> lines(x=Date, y=North.America, col="aquamarine3")
> legend("topleft", legend=c("World", "North America"), col=c("aquamarine4", "aquamarine3"), lty=1, bty="n")
ggplot()設定的是整個畫布的基準,為了讓各線條呈現不同顏色,不在ggplot()中設定而改用geom_line()分別設定線條顏色,再利用scale_color_manual()手動增加圖例。
> p<-ggplot(covid, aes(x=Date))+
+ geom_line(aes(y=World, color="aquamarine4"))+
+ geom_line(aes(y=North.America, color="aquamarine3"))+
+ scale_color_manual(name="", labels=c("World", "North America"), values=c("aquamarine4", "aquamarine3"))+
+ ggtitle("Covid-19 Confirmed Cases")+
+ theme_minimal()
> p
區域圖
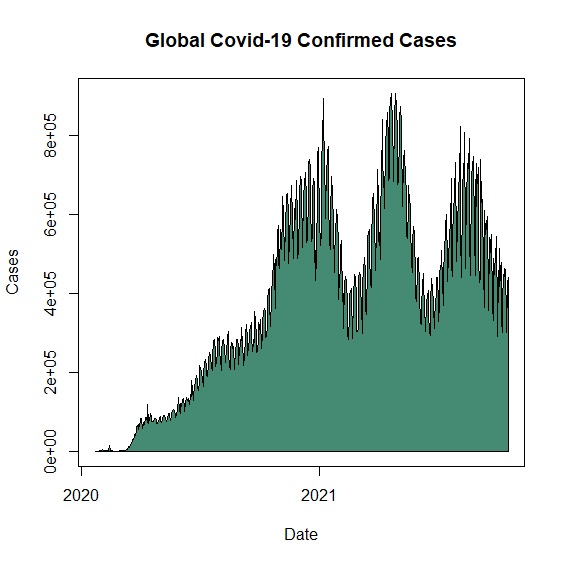
區域圖的概念類似密度圖,是將折線圖下方全部填滿的一種圖形。R Base的區域圖跟密度圖一樣,都是透過polygon()指令來繪製區域,ggplot2則是用geom_area()。設定polygon()的面積區域我們將x軸的Date,設定為從最小到最大,以涵蓋全部的日期。y軸的case從最小再回到最小,以包圍成一個區域。如果y軸第一個變數(起點)與最後一個變數(終點)的數值不是最小,要在頭尾自行增加=0的數值,以確保polygon()能涵蓋整個y軸。
> p<-plot(x=Date, y=World, type="l", xlab="Date", ylab="Cases", main="Global Covid-19 Confirmed Cases", col="aquamarine4")
> polygon(c(min(Date),Date, max(Date)), c(min(World),World,min(World)), col="aquamarine4")
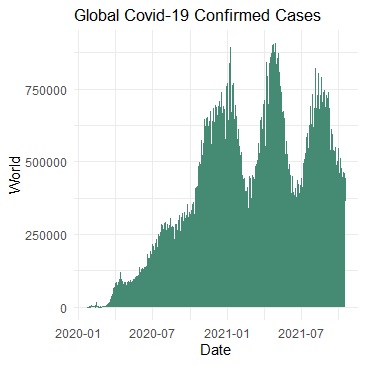
ggplot2用geom_area()設定區域、geom_line()設定線條。
> p<-ggplot(covid, aes(x=Date, y=World))+
+ geom_area(fill="aquamarine4")+
+ geom_line(color="aquamarine4")+
+ ggtitle("Global Covid-19 Confirmed Cases")+
+ theme_minimal()
> p
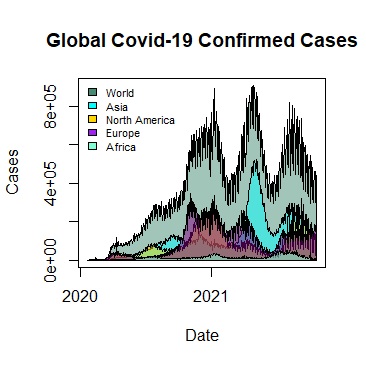
疊合區域圖
多個區域圖的疊合,同樣都是透過polygon()來完成,最後再加上圖例。
> plot(x=Date, y=World, type="l", xlab="Date", ylab="Cases", main="Global Covid-19 Confirmed Cases", col=rgb(0.27,0.55,0.45,0.5))
> polygon(c(min(Date),Date, max(Date)), c(min(World),World,min(World)), col=rgb(0.27,0.55,0.45,0.5))
> polygon(c(min(Date),Date, max(Date)), c(min(Asia),Asia,min(Asia)), col=rgb(0,1,1,0.5))
> polygon(c(min(Date),Date, max(Date)), c(min(North.America),North.America,min(North.America)), col=rgb(1, 0.84, 0, 0.5))
> polygon(c(min(Date),Date, max(Date)), c(min(Europe),Europe,min(Europe)), col=rgb(0.5,0,0.5,0.5))
> polygon(c(min(Date),Date, max(Date)), c(min(Africa),Africa,min(Africa)), col=rgb(0.5,1,0.83,0.5))
> legend("topleft", legend=c("World","Asia","North America","Europe","Africa"), fill=c("aquamarine4","cyan","gold","purple","aquamarine"), bty="n", cex=0.7)
ggplot2的疊合,可以在基本的畫布上ggplot()先完成共同元素的設定後,在用多個geom_area()來疊圖,最後用scale_fill_manual()手動設定圖例。
> p<-ggplot(covid, aes(x=Date))+
+ geom_area(aes(y=World, fill="World"))+
+ geom_area(aes(y=Asia), fill="Asia")+
+ geom_area(aes(y=North.America), fill="North.America")+
+ geom_area(aes(y=Europe), fill="Europe")+
+ geom_area(aes(y=Africa), fill="Africa")+
+ scale_fill_manual(name="", values=c("World"="aquamarine4","Asia"="cyan","North America"="gold","Europe"="purple","Africa"="aquamarine"))+
+ ggtitle("Global Covid-19 Confirmed Cases")+
+ theme_minimal()
> p